

Hier finden Sie wissenschaftliche Publikationen im Zusammenhang mit KI-VesD - dem Forschungsprojekt zur Entwicklung einer KI-gesteuerten Plattformtechnologie für die Bewertung der Proteaseaktivität in Plasmavesikeln.
Die wissenschaftlichen Publikationen auf dieser Seite dokumentieren den Fortschritt und die Ergebnisse des KI-VesD - Projekts. Die Veröffentlichungen beschreiben die Methoden und Technologien, die im Projekt eingesetzt wurden, sowie die Ergebnisse von Tests und Experimenten.
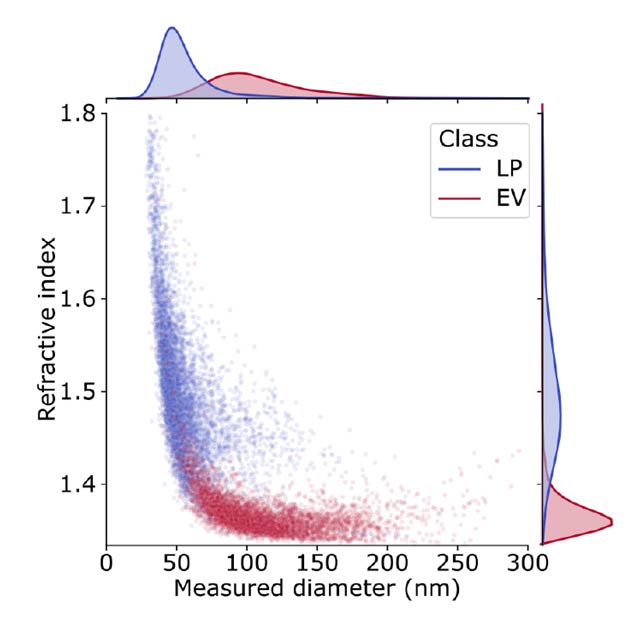
Accurate quantification of EVs has diagnostic value but is a challenge, due to their nanometer size range. The lab of Vahid Sandoghdar at the Max Planck Institute for the Science of Light has developed interferometric Nanoparticle Tracking Analysis (iNTA), a highly sensitive technology capable of analyzing nanoparticles in solution. In a recent collaboration, we have shown that iNTA is able to distinguish EVs from other plasma-derived particles in a label-free manner, down to a size of 40 nm. This outperforms all existing instruments on the market, as well as other lab-developed approaches such as high-resolution flow cytometry.
Veröffentlicht von:
Anna D. Kashkanova, Martin Blessing, Marie Reischke, Jan-Ole Baur, Andreas S. Baur, Vahid Sandoghdar, Jan Van Deun
Vollständige Veröffentlichung lesen:
https://doi.org/10.1002/jev2.12348
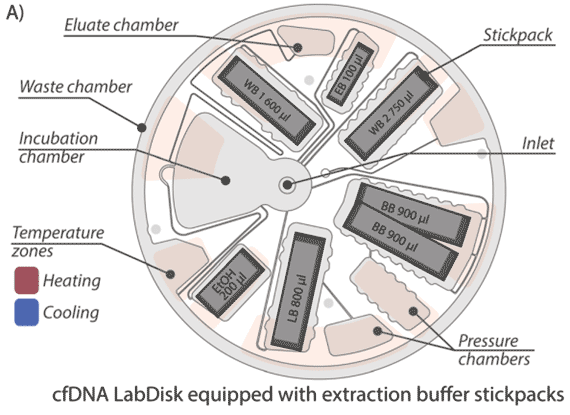

Diese Publikation demonstriert das Potenzial der LabDisk-Mikrofluidikplattform für die Biomarkerextraktion (Flüssigbiopsie) am Beispiel von zellfreier DNA (cfDNA). cfDNA, die von Zellen in den Blutkreislauf abgegeben wird, kann spezifische Informationen enthalten, z.B. genetische Informationen über Tumore. Diese Arbeit demonstriert ein neues Konzept für eine automatisierte cfDNA-Extraktion mit Nanobeads als fester Phase in einer zentrifugalen mikrofluidischen LabDisk. Durch eine Kombination von Zentrifugal- und Magnetkräften halten wir die Nanobeads in einer Inkubationskammer. Die cfDNA aus der Plasmaprobe wird an die Nanobeads gebunden, gewaschen und anschließend eluiert und für genetische Analysen zur Verfügung gestellt.
Veröffentlicht von:
Schlenker F, Juelg P, Lüddecke J, Paust N, Zengerle R, Hutzenlaub T
Vollständige Veröffentlichung lesen:
https://doi.org/10.1039/D2AN01820A
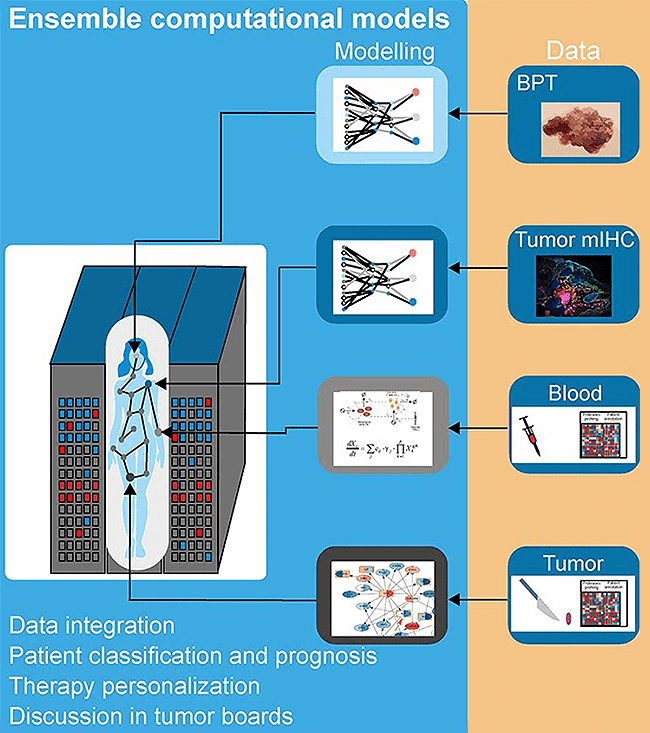
Melanoma is a type of skin cancer that is relatively common in populations of European origin. When not detected in early phases, it gets metastatic. Metastatic melanoma was untreatable until a few years ago. Nowadays, novel cancer immunotherapies are effective for half of metastatic melanoma patients. However, many still do not respond to the therapy, and others develop severe side effects.
We hypothesise that melanoma is ideal for developing a modern medicine based on molecular analysis and artificial intelligence (AI)-based computer models. There is a medical need for better diagnostics and treatment. Also, the skin is an organ easy to access for many technologies used to generate the quantitative data necessary for AI models like advanced imaging. In this work, written by a multidisciplinary team of clinicians, engineers, physicists, and biomedical researchers, we discuss combining AI models and patient molecular data to improve diagnostics. We focus on liquid biopsies like the profiling of blood serum and extracellular vesicles.
Veröffentlicht von:
Vera J, Lai X, Baur A, Erdmann M, Gupta S, Guttà C, Heinzerling L, Heppt MV, Kazmierczak PM, Kunz M, Lischer C, Pützer BM, Rehm M, Ostalecki C, Retzlaff J, Witt S, Wolkenhauer O, Berking C.
Vollständige Veröffentlichung lesen:
https://doi.org/10.1093/bib/bbac433
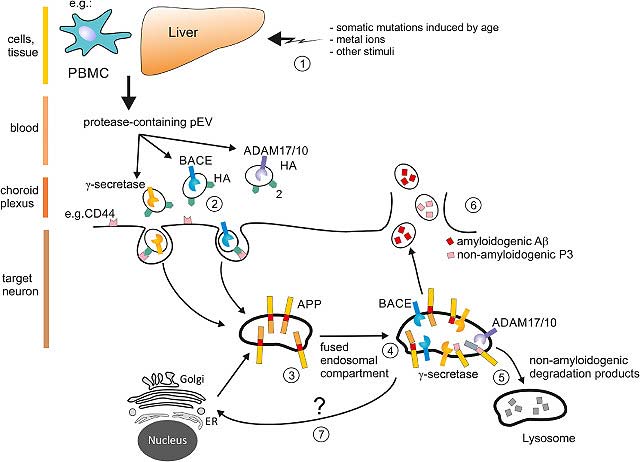
Diese Publikation zeigt erstmals wie bei der Alzheimererkrankung Protease-haltige Vesikel genau dort das Gehirn erreichen, wo die Alzheimer Kranken den Ausgang nimmt, nämlich dem Hippocampus. Wir nehmen an, dass die mitgebrachten Proteasen den Hippocampus nachhaltig schädigen und eben jene Peptide produzieren, die zum Kennzeichen der Alzheimererkrankung wurden (sogenannte Plaques). Nachdem die Konzentration dieser Vesikel bei Alzheimer-Patienten bis um das hundertfache gesteigert ist, ist der Schaden besonders, und nimmt mit fortschreitender Erkrankung zu. Den Stand unserer Forschung zu diesem Thema haben wir in einem Artikel in EBioMedicine dargestellt. Dazu gehört ein Begleit-Kommentar eines Experten aus dem Feld.
Veröffentlicht von:
Jung-Hyun Lee, Christian Ostalecki, Timo Oberstein, Stefan Schierer, Elisabeth Zinser, Martin Eberhardt, Katja Blume, Bianca Plosnita, Lena Stich, Heiko Bruns, Roland Coras, Julio Vera-Gonzales, Manuel Maler, Andreas S. Baur
Vollständige Veröffentlichung lesen:
https://doi.org/10.1016/j.ebiom.2022.103903
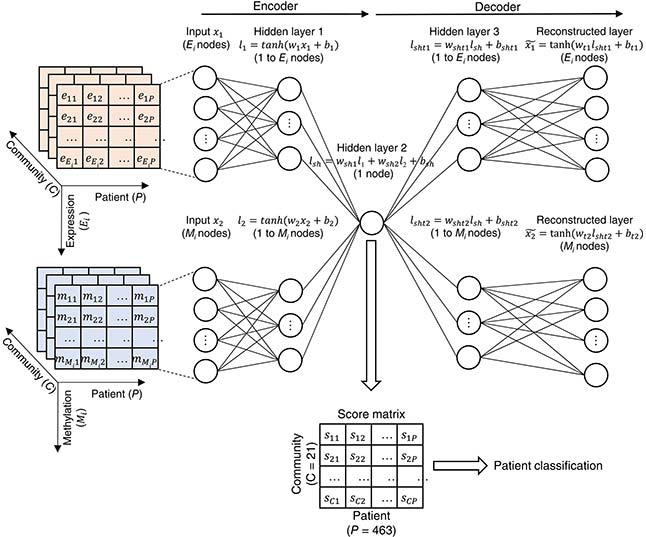
There is a medical need to develop better diagnostics tools for melanoma. Combining molecular analysis and artificial intelligence is the way to achieve it. Here, we integrate patients’ tumour genomics data and deep learning to classify melanoma patients. Deep learning models are large and advanced AI models behind recent breakthroughs like ChatGPT. A central feature of the method is that we do not consider genes individually but as a part of networks of cancer genes influencing each other to make the tumor worse. This way, one can make a better patient classification. Also, one can understand the biological processes behind cancer aggressiveness. Understanding these processes is the first step to developing new, more effective therapies.
Veröffentlicht von:
Lai X, Zhou J, Wessely A, Heppt M, Maier A, Berking C, Vera J, Zhang L.
Vollständige Veröffentlichung lesen:
https://doi.org/10.1002/ijc.33860
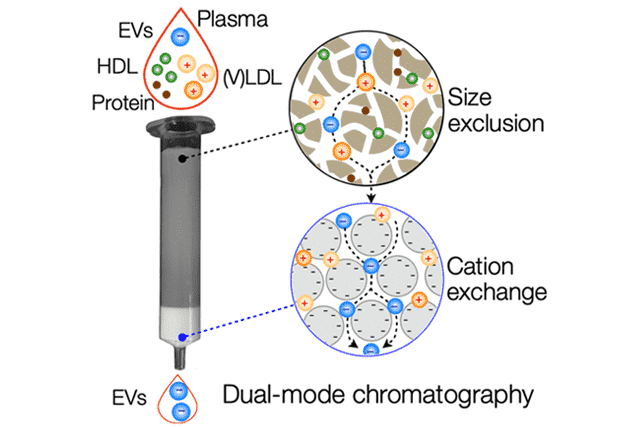
Purifying extracellular vesicles (EVs) from complex biological fluids, such as blood plasma, is a critical step for their clinical application. Plasma lipoproteins are a significant confounding factor as they outnumber EVs by more than 10000-fold. Here, we developed a new separation approach, known as dual-mode chromatography (DMC), which can quickly and reliably enrich EVs from plasma, depleting lipoproteins by well over 99%.
Veröffentlicht von:
Jan Van Deun, Ala Jo, Huiyan Li, Hsing-Ying Lin, Ralph Weissleder, Hyungsoon Im, Hakho Lee
Vollständige Veröffentlichung lesen:
https://doi.org/10.1002/adbi.201900310
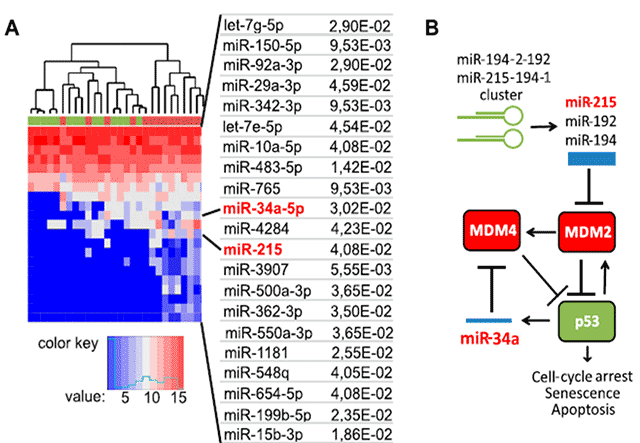
Diese Publikation zeigt erstmals wie vom Körper produzierte extrazelluläre Vesikel Tumorzellen in ihrer Proliferation hemmen. Dies lieferte eine Erklärung warum diese Vesikel bei Tumorpatienten kontinuierlich produziert werden, wahrscheinlich ein Leben lang, und Informationen zum Stand Erkrankungen liefern können. Diese Befunde lieferten ein Rational für die Entwicklung unseres Testsystems.
Weiter zeigt Publikation erstmals, dass die Höhe von Protease-Aktivität in pEV mit dem klinischen Stadium einer Krebserkrankung, hier die Melanom, korreliert. Je weiter fortgeschritten das klinische Stadium, desto höher war die durchschnittliche Protease-Aktivität. Das Besondere an diesem Befund war die Tatsache, dass die Patienten bereits operiert waren, d. h. der sichtbare Tumor war entfernt worden. Dies bedeutete dass das Testsystem sogar die Restmenge an Tumorzellen erkennen konnte, selbst wenn die Operation Jahre zurücklag, und der Tumor ursprünglich sehr klein war.
Veröffentlicht von:
Lee JH, Dindorf J, Eberhardt M, Lai X, Ostalecki C, Koliha N, et al., and Baur AS
Vollständige Veröffentlichung lesen:
https://doi.org/10.26508/lsa.201800205
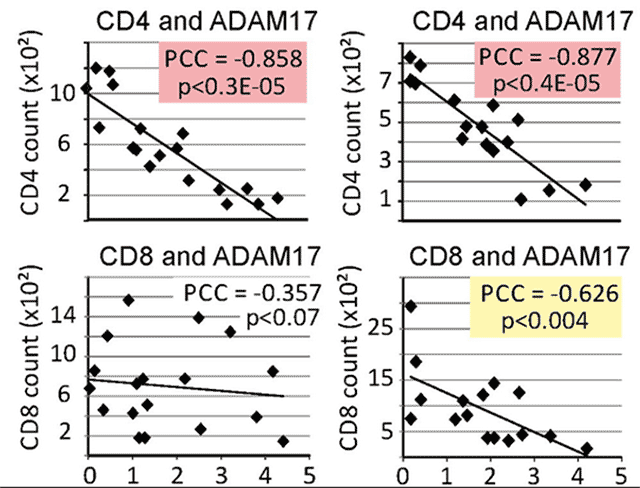
In diesem Paper wurde erstmals ein Zusammenhang gefunden zwischen der Protease-Aktivität in pEV und dem klinischen Stadium einer Erkrankung, nämlich der chronischen HIV infection. Die Proteaseaktivität von ADAM17 korrelierte hochsignifikant mit den Zahlen von CD4 Helferzellen bei HIV-Patienten. Je niedriger die CD4 T-Zellen, desto höher war die Aktivität von ADAM17 in pEV.
Veröffentlicht von:
Lee, J-H, Schierer,S., Blume, K., Dindorf, J., Wittki, S., Xiang, W., et al. and Baur, AS.
Vollständige Veröffentlichung lesen:
https://doi.org/10.1016%2Fj.ebiom.2016.03.004
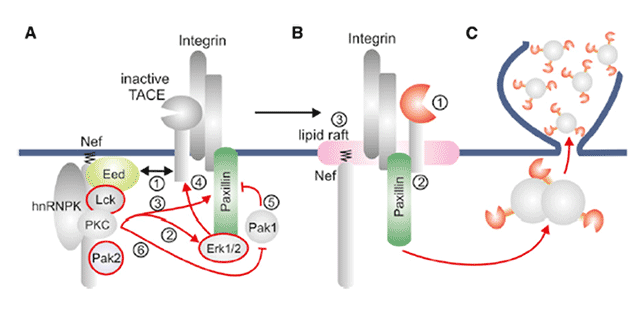
Diese Publikation beschreibt den molekularen Mechanismus der zur Verpackung von Proteasen in extrazellulären Vesikel führt. Diese Publikation wurde erstmals unser neues Testsystem angewendet, welches kurz vor der Publikation des Artikels patentiert wurde. Der darin gefunden Mechanismus war die Grundlage für die Entwicklung des Testsystems.
Veröffentlicht von:
Lee, J.H., et al. and Baur, A.S.
Vollständige Veröffentlichung lesen:
https://doi.org/10.1016/j.molcel.2012.12.004
